# Instalar paquetes si no los tienes
# install.packages("tidyverse")
# install.packages("haven")
# install.packages("nortest") # Para la prueba de Lilliefors (K-S)
# install.packages("car") # Para el Test de Levene
library(tidyverse)
library(haven)
library(nortest)
library(car)
library(expss)1 Exploración de Datos y Validación de Supuestos en R
1.1 Introducción
Bienvenido a la guía fundamental de análisis de datos en R. Este documento, que hemos llamado “Punto 0”, cubre el paso más crucial de cualquier proyecto de investigación: la exploración inicial de los datos y la validación de los supuestos estadísticos. Antes de poder aplicar técnicas de inferencia como el test t o el ANOVA, debemos entender a fondo nuestras variables y comprobar si cumplen con los requisitos de las pruebas paramétricas.
A lo largo de este documento, replicaremos los procedimientos que tradicionalmente se realizan en SPSS, demostrando la sintaxis y las capacidades de R y RStudio. Nuestro objetivo es construir una base sólida para los análisis más avanzados que realizaremos en las siguientes sesiones.
Primero, cargamos los paquetes que necesitaremos. tidyverse es una colección de paquetes para la manipulación y visualización de datos, haven nos permite leer ficheros de SPSS (.sav), y nortest contiene la prueba de Kolmogorov-Smirnov con la corrección de Lilliefors.
1.2 1. Carga y Exploración Inicial del Fichero de Datos
En SPSS, abriríamos un fichero .sav a través del menú. En R, podemos hacer lo mismo de forma programática, lo que garantiza la reproducibilidad de nuestro análisis. Usaremos el fichero gssnet2.sav que se menciona en el material del curso.
# Cargar el fichero de datos de SPSS
# Asegúrate de que el fichero "gssnet2.sav" esté en tu proyecto de RStudio
# o proporciona la ruta completa al archivo.
# Por ejemplo: datos <- read_sav("C:/ruta/a/tus/datos/gssnet2.sav")
# Para este ejemplo, si no tienes el fichero, crearemos datos simulados
# que se parezcan a los del ejemplo (edad y horas de uso de internet por sexo)
# ¡Cuando uses tus datos, reemplaza este bloque con la línea de read_sav!
datos <- read_sav('data/gssnet1.sav')
# Vistazo inicial a los datos (equivalente a la "Vista de Datos" de SPSS)
glimpse(datos)Rows: 984
Columns: 10
$ age <dbl+lbl> 30, 39, 72, 41, 24, 23, 27, 34, 45, 51, 46, 56, 46, 23, 5…
$ educ <dbl+lbl> 11, 9, 10, 13, 12, 12, 12, 10, 11, 12, 12, 6, 10, 10, 1…
$ usecomp <dbl+lbl> 0, 0, 0, 0, 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 0, 0, …
$ usenet <dbl+lbl> 0, 0, 0, 0, 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 0, 0, …
$ usemail <dbl+lbl> 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0, 1, 1, 0, 0, 0, …
$ emailhrs <dbl+lbl> NA, NA, NA, NA, 1.00…
$ webhrs <dbl+lbl> NA, NA, NA, NA, 2.0, NA, 0.5, NA, NA, N…
$ nethrs <dbl+lbl> NA, NA, NA, NA, 3.000000, …
$ netcat <dbl+lbl> NA, NA, NA, NA, 1, NA, 1, NA, NA, NA, NA, NA, NA, NA, …
$ region <dbl+lbl> 5, 7, 3, 3, 6, 5, 7, 4, 3, 5, 6, 7, 7, 7, 7, 2, 3, 3, 3, …# Resumen estadístico rápido de todas las variables
summary(datos) age educ usecomp usenet
Min. :18.00 Min. : 0.00 Min. :0.0000 Min. :0.0000
1st Qu.:33.00 1st Qu.:12.00 1st Qu.:0.0000 1st Qu.:0.0000
Median :45.00 Median :14.00 Median :1.0000 Median :1.0000
Mean :46.25 Mean :13.74 Mean :0.7393 Mean :0.7393
3rd Qu.:58.00 3rd Qu.:16.00 3rd Qu.:1.0000 3rd Qu.:1.0000
Max. :89.00 Max. :20.00 Max. :1.0000 Max. :1.0000
NA's :4 NA's :6 NA's :6
usemail emailhrs webhrs nethrs
Min. :0.0000 Min. : 0.01667 Min. : 0.000 Min. : 0.08333
1st Qu.:0.0000 1st Qu.: 1.00000 1st Qu.: 1.000 1st Qu.: 3.00000
Median :1.0000 Median : 2.00000 Median : 4.000 Median : 7.00000
Mean :0.6399 Mean : 6.09360 Mean : 6.732 Mean : 12.82531
3rd Qu.:1.0000 3rd Qu.: 7.00000 3rd Qu.: 8.000 3rd Qu.: 15.50000
Max. :1.0000 Max. :50.00000 Max. :70.000 Max. :118.00000
NA's :12 NA's :367 NA's :367 NA's :367
netcat region
Min. :1.000 Min. :1.000
1st Qu.:1.000 1st Qu.:3.000
Median :2.000 Median :5.000
Mean :2.361 Mean :5.117
3rd Qu.:3.000 3rd Qu.:7.000
Max. :4.000 Max. :9.000
NA's :367 1.3 2. Tablas de Frecuencias y Estadísticos Descriptivos
Esta sección replica los procedimientos de Analizar > Estadísticos Descriptivos > Frecuencias y Descriptivos en SPSS.
1.3.1 2.1. Variables Categóricas: Tabla de Frecuencias
Para variables como sex, queremos contar cuántos casos hay en cada categoría.
# Tabla de frecuencias para la variable 'sex'
frecuencias_sexo <- fre(datos$age)
# Mostramos la tabla
knitr::kable(frecuencias_sexo, caption = "Tabla de Frecuencias para Género")| Age of respondent | Count | Valid percent | Percent | Responses, % | Cumulative responses, % |
|---|---|---|---|---|---|
| 18 | 10 | 1.0204082 | 1.0162602 | 1.0204082 | 1.020408 |
| 19 | 10 | 1.0204082 | 1.0162602 | 1.0204082 | 2.040816 |
| 20 | 6 | 0.6122449 | 0.6097561 | 0.6122449 | 2.653061 |
| 21 | 13 | 1.3265306 | 1.3211382 | 1.3265306 | 3.979592 |
| 22 | 9 | 0.9183673 | 0.9146341 | 0.9183673 | 4.897959 |
| 23 | 18 | 1.8367347 | 1.8292683 | 1.8367347 | 6.734694 |
| 24 | 26 | 2.6530612 | 2.6422764 | 2.6530612 | 9.387755 |
| 25 | 21 | 2.1428571 | 2.1341463 | 2.1428571 | 11.530612 |
| 26 | 20 | 2.0408163 | 2.0325203 | 2.0408163 | 13.571429 |
| 27 | 17 | 1.7346939 | 1.7276423 | 1.7346939 | 15.306122 |
| 28 | 22 | 2.2448980 | 2.2357724 | 2.2448980 | 17.551020 |
| 29 | 12 | 1.2244898 | 1.2195122 | 1.2244898 | 18.775510 |
| 30 | 19 | 1.9387755 | 1.9308943 | 1.9387755 | 20.714286 |
| 31 | 17 | 1.7346939 | 1.7276423 | 1.7346939 | 22.448980 |
| 32 | 19 | 1.9387755 | 1.9308943 | 1.9387755 | 24.387755 |
| 33 | 23 | 2.3469388 | 2.3373984 | 2.3469388 | 26.734694 |
| 34 | 24 | 2.4489796 | 2.4390244 | 2.4489796 | 29.183674 |
| 35 | 24 | 2.4489796 | 2.4390244 | 2.4489796 | 31.632653 |
| 36 | 12 | 1.2244898 | 1.2195122 | 1.2244898 | 32.857143 |
| 37 | 13 | 1.3265306 | 1.3211382 | 1.3265306 | 34.183673 |
| 38 | 22 | 2.2448980 | 2.2357724 | 2.2448980 | 36.428571 |
| 39 | 13 | 1.3265306 | 1.3211382 | 1.3265306 | 37.755102 |
| 40 | 27 | 2.7551020 | 2.7439024 | 2.7551020 | 40.510204 |
| 41 | 17 | 1.7346939 | 1.7276423 | 1.7346939 | 42.244898 |
| 42 | 17 | 1.7346939 | 1.7276423 | 1.7346939 | 43.979592 |
| 43 | 19 | 1.9387755 | 1.9308943 | 1.9387755 | 45.918367 |
| 44 | 24 | 2.4489796 | 2.4390244 | 2.4489796 | 48.367347 |
| 45 | 19 | 1.9387755 | 1.9308943 | 1.9387755 | 50.306122 |
| 46 | 26 | 2.6530612 | 2.6422764 | 2.6530612 | 52.959184 |
| 47 | 21 | 2.1428571 | 2.1341463 | 2.1428571 | 55.102041 |
| 48 | 24 | 2.4489796 | 2.4390244 | 2.4489796 | 57.551020 |
| 49 | 26 | 2.6530612 | 2.6422764 | 2.6530612 | 60.204082 |
| 50 | 26 | 2.6530612 | 2.6422764 | 2.6530612 | 62.857143 |
| 51 | 18 | 1.8367347 | 1.8292683 | 1.8367347 | 64.693878 |
| 52 | 18 | 1.8367347 | 1.8292683 | 1.8367347 | 66.530612 |
| 53 | 10 | 1.0204082 | 1.0162602 | 1.0204082 | 67.551020 |
| 54 | 17 | 1.7346939 | 1.7276423 | 1.7346939 | 69.285714 |
| 55 | 15 | 1.5306122 | 1.5243902 | 1.5306122 | 70.816327 |
| 56 | 21 | 2.1428571 | 2.1341463 | 2.1428571 | 72.959184 |
| 57 | 18 | 1.8367347 | 1.8292683 | 1.8367347 | 74.795918 |
| 58 | 13 | 1.3265306 | 1.3211382 | 1.3265306 | 76.122449 |
| 59 | 14 | 1.4285714 | 1.4227642 | 1.4285714 | 77.551020 |
| 60 | 17 | 1.7346939 | 1.7276423 | 1.7346939 | 79.285714 |
| 61 | 14 | 1.4285714 | 1.4227642 | 1.4285714 | 80.714286 |
| 62 | 19 | 1.9387755 | 1.9308943 | 1.9387755 | 82.653061 |
| 63 | 9 | 0.9183673 | 0.9146341 | 0.9183673 | 83.571429 |
| 64 | 10 | 1.0204082 | 1.0162602 | 1.0204082 | 84.591837 |
| 65 | 7 | 0.7142857 | 0.7113821 | 0.7142857 | 85.306122 |
| 66 | 11 | 1.1224490 | 1.1178862 | 1.1224490 | 86.428571 |
| 67 | 12 | 1.2244898 | 1.2195122 | 1.2244898 | 87.653061 |
| 68 | 7 | 0.7142857 | 0.7113821 | 0.7142857 | 88.367347 |
| 69 | 8 | 0.8163265 | 0.8130081 | 0.8163265 | 89.183673 |
| 70 | 11 | 1.1224490 | 1.1178862 | 1.1224490 | 90.306122 |
| 71 | 8 | 0.8163265 | 0.8130081 | 0.8163265 | 91.122449 |
| 72 | 8 | 0.8163265 | 0.8130081 | 0.8163265 | 91.938776 |
| 73 | 7 | 0.7142857 | 0.7113821 | 0.7142857 | 92.653061 |
| 74 | 11 | 1.1224490 | 1.1178862 | 1.1224490 | 93.775510 |
| 75 | 7 | 0.7142857 | 0.7113821 | 0.7142857 | 94.489796 |
| 76 | 5 | 0.5102041 | 0.5081301 | 0.5102041 | 95.000000 |
| 77 | 2 | 0.2040816 | 0.2032520 | 0.2040816 | 95.204082 |
| 78 | 8 | 0.8163265 | 0.8130081 | 0.8163265 | 96.020408 |
| 79 | 7 | 0.7142857 | 0.7113821 | 0.7142857 | 96.734694 |
| 80 | 2 | 0.2040816 | 0.2032520 | 0.2040816 | 96.938776 |
| 81 | 4 | 0.4081633 | 0.4065041 | 0.4081633 | 97.346939 |
| 82 | 4 | 0.4081633 | 0.4065041 | 0.4081633 | 97.755102 |
| 83 | 4 | 0.4081633 | 0.4065041 | 0.4081633 | 98.163265 |
| 84 | 3 | 0.3061224 | 0.3048780 | 0.3061224 | 98.469388 |
| 85 | 4 | 0.4081633 | 0.4065041 | 0.4081633 | 98.877551 |
| 86 | 3 | 0.3061224 | 0.3048780 | 0.3061224 | 99.183673 |
| 87 | 1 | 0.1020408 | 0.1016260 | 0.1020408 | 99.285714 |
| 88 | 2 | 0.2040816 | 0.2032520 | 0.2040816 | 99.489796 |
| 89 | 5 | 0.5102041 | 0.5081301 | 0.5102041 | 100.000000 |
| #Total | 980 | 100.0000000 | 99.5934959 | 100.0000000 | NA |
| 4 | NA | 0.4065041 | NA | NA |
1.3.2 2.2. Variables Numéricas: Estadísticos Descriptivos
Para una variable continua como age, nos interesan medidas de tendencia central, dispersión y forma.
# Cálculo de descriptivos para la variable 'age'
descriptivos_edad <- datos %>%
summarise(
N_Validos = n(),
Media = mean(age, na.rm = TRUE),
Mediana = median(age, na.rm = TRUE),
Desv_Estandar = sd(age, na.rm = TRUE),
Minimo = min(age, na.rm = TRUE),
Maximo = max(age, na.rm = TRUE),
Rango = Maximo - Minimo,
Asimetria = moments::skewness(age, na.rm = TRUE),
Curtosis = moments::kurtosis(age, na.rm = TRUE) - 3 # Se resta 3 para obtener la curtosis "excesiva"
)
# Mostramos la tabla de descriptivos
knitr::kable(descriptivos_edad, caption = "Estadísticos Descriptivos para Edad")| N_Validos | Media | Mediana | Desv_Estandar | Minimo | Maximo | Rango | Asimetria | Curtosis |
|---|---|---|---|---|---|---|---|---|
| 984 | 46.25408 | 45 | 16.72287 | 18 | 89 | 71 | 0.3882235 | -0.5896465 |
Interpretación de Asimetría y Curtosis: - Asimetría: Un valor cercano a 0 indica simetría. Valores > 0 indican una cola larga a la derecha (sesgo positivo); valores < 0 indican una cola larga a la izquierda (sesgo negativo). - Curtosis (Excesiva): Mide qué tan “puntiaguda” es la distribución y cómo de pesadas son sus colas. Un valor cercano a 0 es similar a una normal (mesocúrtica). Un valor > 0 indica una forma más puntiaguda con colas pesadas (leptocúrtica). Un valor < 0 indica una forma más aplanada con colas ligeras (platicúrtica).
1.4 3. Comprobación de la Normalidad
Este es un supuesto clave para muchas pruebas paramétricas. Lo evaluaremos de dos formas: gráfica y estadística, replicando el menú Explorar de SPSS.
Hipótesis en las pruebas de normalidad: - Hipótesis Nula (H₀): Los datos provienen de una población con distribución normal. - Hipótesis Alternativa (Hₐ): Los datos NO provienen de una población con distribución normal.
Si el p-valor es < 0.05, rechazamos la H₀ y concluimos que no hay evidencia de normalidad.
1.4.1 3.1. Métodos Gráficos
Los gráficos son a menudo más informativos que las pruebas formales, especialmente con muestras grandes.
# Histograma con curva de densidad normal superpuesta
ggplot(datos, aes(x = age)) +
geom_histogram(aes(y = ..density..), bins = 20, fill = "lightblue", color = "black") +
stat_function(fun = dnorm, args = list(mean = mean(datos$age), sd = sd(datos$age)), color = "red", size = 1) +
labs(title = "Histograma de Edad con Curva Normal", x = "Edad", y = "Densidad") +
theme_minimal()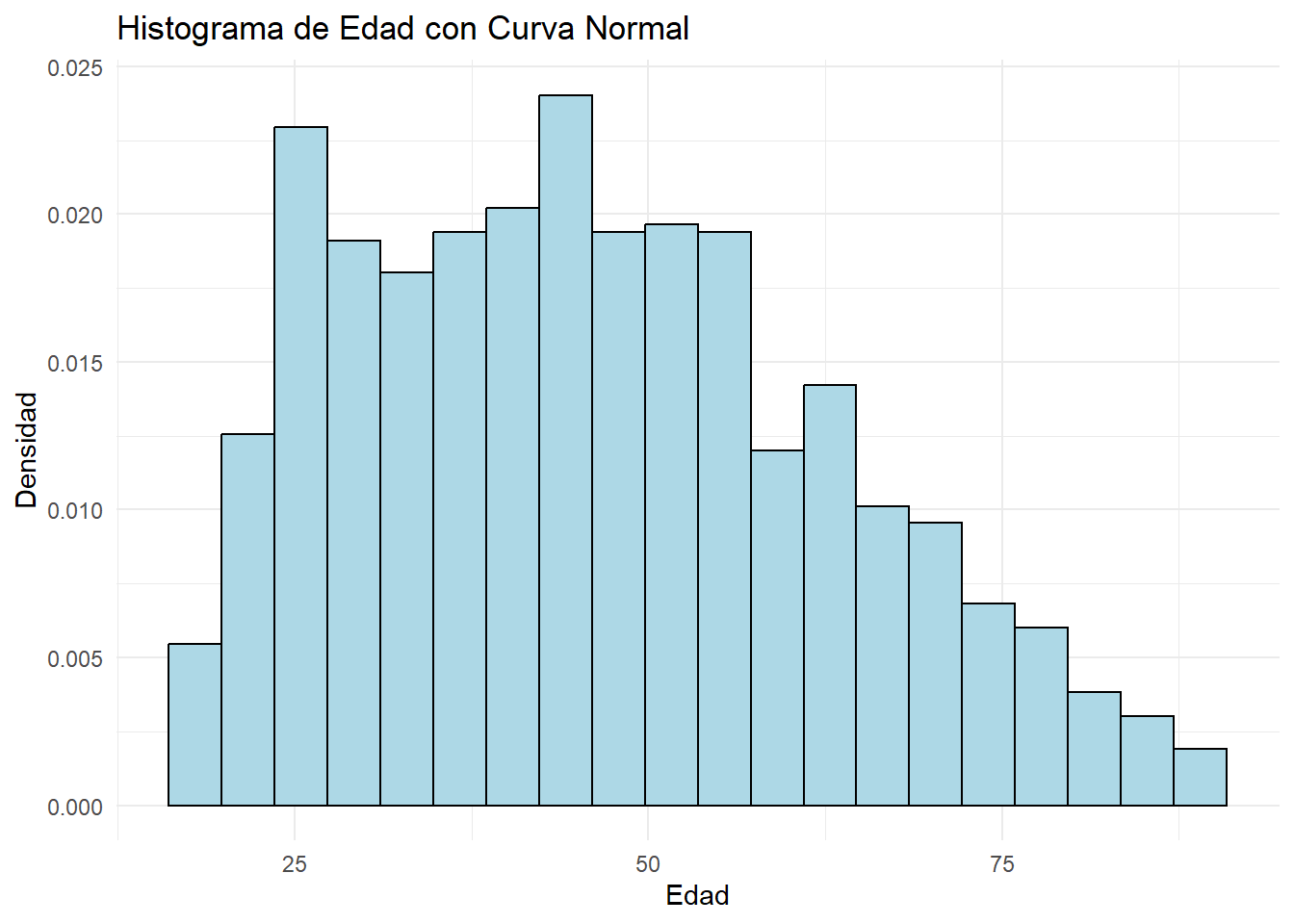
# Gráfico Q-Q (Quantile-Quantile)
ggplot(datos, aes(sample = age)) +
stat_qq() +
stat_qq_line(color = "blue") +
labs(title = "Gráfico Q-Q para la Variable Edad", x = "Cuantiles Teóricos (Normal)", y = "Cuantiles de la Muestra") +
theme_minimal()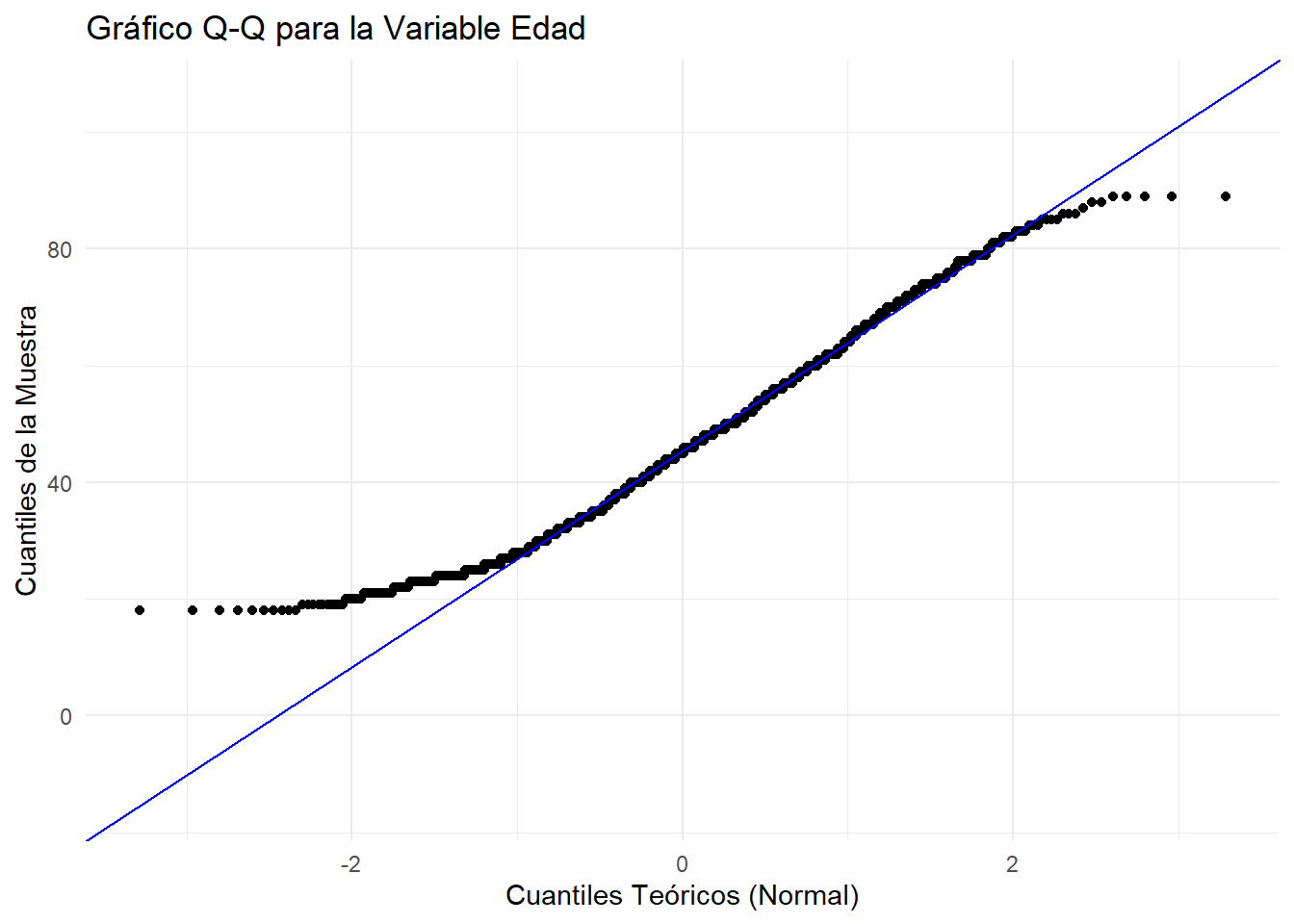
Interpretación Gráfica: - Histograma: Comparamos visualmente la forma de las barras con la curva normal teórica (roja). - Gráfico Q-Q: Si los puntos se ajustan estrechamente a la línea diagonal, es una fuerte indicación de normalidad. Desviaciones sistemáticas (en forma de “S” o “banana”) sugieren no-normalidad.
1.4.2 3.2. Pruebas Estadísticas
1.4.2.1 a) Prueba de Shapiro-Wilk
Es la prueba más potente y recomendada para la normalidad, especialmente con muestras de menos de 2000-5000 casos.
# Realizamos la prueba de Shapiro-Wilk
shapiro_test_resultado <- shapiro.test(datos$age)
print(shapiro_test_resultado)
Shapiro-Wilk normality test
data: datos$age
W = 0.97233, p-value = 1.033e-12Interpretación: Observamos el p-value. Si es menor a 0.05, rechazamos la normalidad.
1.4.2.2 b) Prueba de Kolmogorov-Smirnov con corrección de Lilliefors
Es una alternativa, aunque generalmente menos potente que Shapiro-Wilk para probar la normalidad.
# Realizamos la prueba de K-S con corrección de Lilliefors
lillie_test_resultado <- lillie.test(datos$age)
print(lillie_test_resultado)
Lilliefors (Kolmogorov-Smirnov) normality test
data: datos$age
D = 0.065845, p-value = 8.869e-11Interpretación: La lógica es la misma. Si el p-value es menor a 0.05, rechazamos la normalidad.
1.5 4. Comprobación de la Homoscedasticidad
Esta es la prueba formal para la igualdad de varianzas. Al igual que en la documentación usamos otro fichero, hatco.sav.
La homoscedasticidad, o igualdad de varianzas, es un supuesto clave para pruebas que comparan grupos (como el test t para muestras independientes o el ANOVA). Significa que la dispersión de la variable continua es la misma en todos los grupos.
Hipótesis en el Test de Levene: - Hipótesis Nula (H₀): Las varianzas de los grupos son iguales (hay homoscedasticidad). - Hipótesis Alternativa (Hₐ): Al menos una varianza es diferente (hay heteroscedasticidad).
Si el p-valor es < 0.05, rechazamos la H₀ y concluimos que las varianzas no son homogéneas.
1.5.1 Ejemplo: ¿La dispersión de la valoración en la rapidez del servicio y/o en el nivel de servicio es la misma para las empresas grandes y pequeñas?
1.5.2 4.1. Método Gráfico
Los diagramas de caja (boxplots) son la mejor herramienta visual para comparar la dispersión entre grupos.
datos <- read_sav('data/hatco.sav')
# Diagramas de caja
ggplot(datos, aes(x = X8, y = X1 , fill = X8)) +
geom_boxplot() +
labs(
title = "Dispersión de Valoración por Tamaño de empresa",
x = "Tamaño empresa",
y = "Valoración de la rapidez de servicio"
) +
theme_minimal() +
theme(legend.position = "none")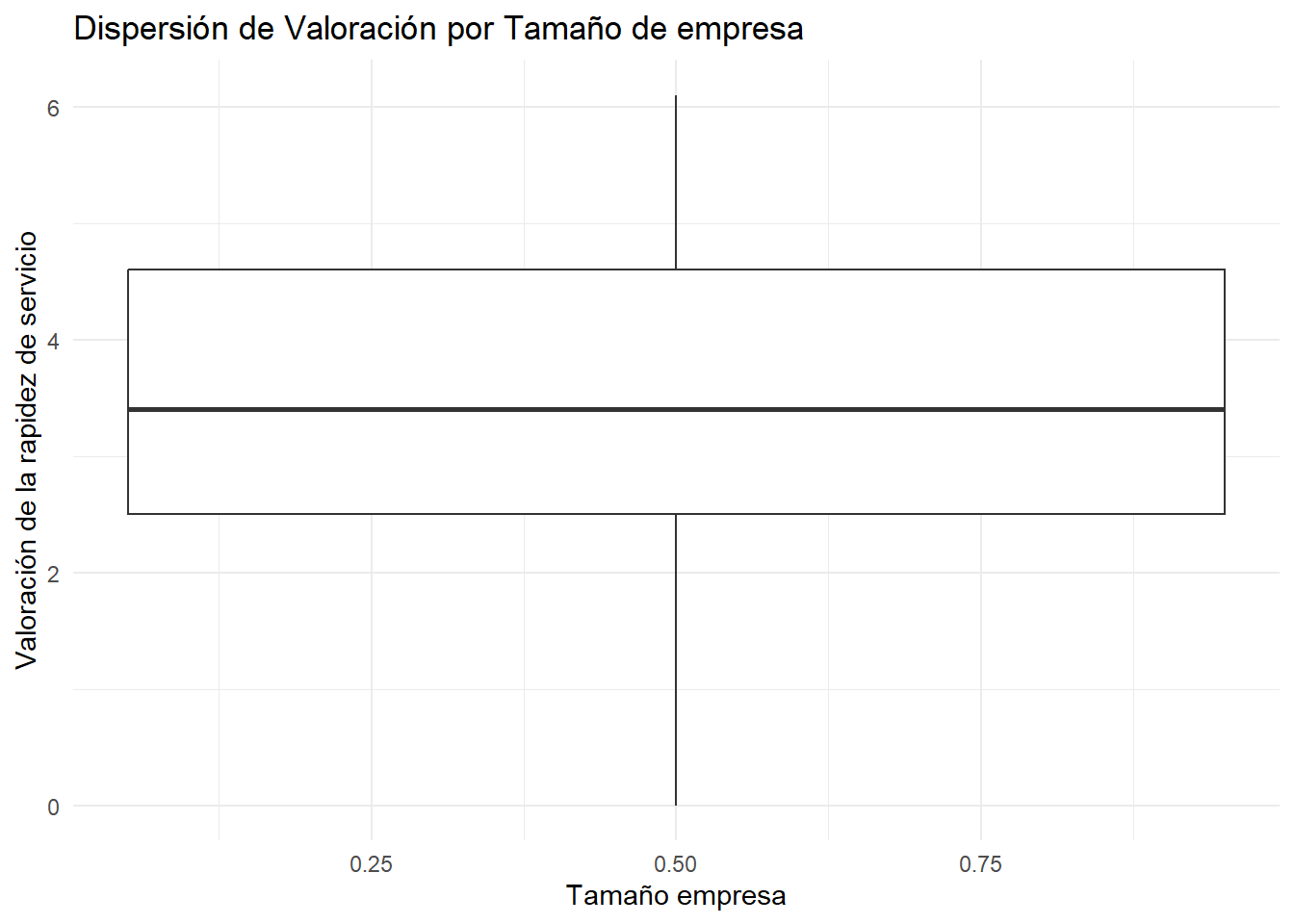
ggplot(datos, aes(x = X8, y = X5, fill = X8)) +
geom_boxplot() +
labs(
title = "Dispersión de Valoración por Tamaño de empresa",
x = "Tamaño empresa",
y = "Valoración del nivel de servicio"
) +
theme_minimal() +
theme(legend.position = "none")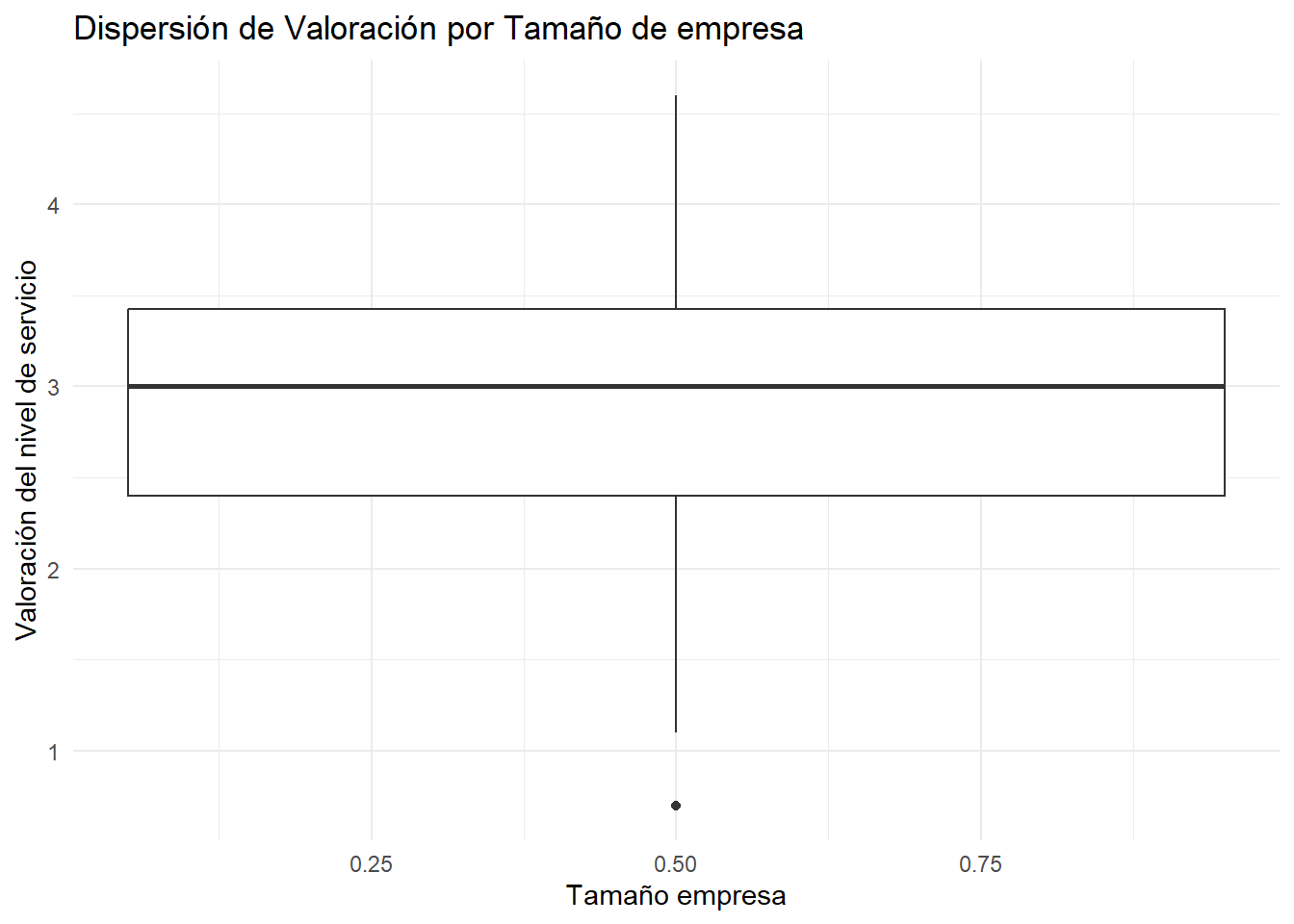
Interpretación Gráfica: Comparamos la altura de las cajas (que representa el Rango Intercuartílico). Si las cajas tienen alturas muy diferentes, podría ser un indicio de heteroscedasticidad.
1.5.3 4.2. Prueba Estadística: Test de Levene
# Realizamos el Test de Levene
levene_test_resultado1 <- leveneTest(X1 ~ factor(X8), data = datos)
levene_test_resultado2 <- leveneTest(X5 ~ factor(X8), data = datos)
print(levene_test_resultado1)Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 1 0.993 0.3215
98 print(levene_test_resultado2)Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 1 6.7411 0.01087 *
98
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Interpretación: Observamos el p-valor (Pr(>F)). Si es menor a 0.05, concluimos que las varianzas son significativamente diferentes. Si es mayor a 0.05, no tenemos evidencia para rechazar la H₀ y podemos asumir homoscedasticidad.
1.6 Conclusión
Hemos completado el primer paso (“Punto 0”) de nuestro viaje en el análisis de datos. Ahora sabemos cómo cargar datos, obtener descriptivos y, lo más importante, cómo validar los supuestos de normalidad y homoscedasticidad en R. Esta base nos permitirá elegir con confianza entre pruebas paramétricas y no paramétricas en las próximas sesiones. ```